2023年7月28日,来自斯坦福大学医学院分子和细胞生理学以及结构生物学教授K. Christopher Garcia团队在Science上发表文章Deploying synthetic coevolution and machine learning to engineer protein-protein interactions。
文章构建了一个蛋白质-蛋白质共进化平台,可以从复杂的文库中分离相互作用的蛋白质。此外将合成共同进化与机器学习相结合,可以预测重构的蛋白质-蛋白质界面。

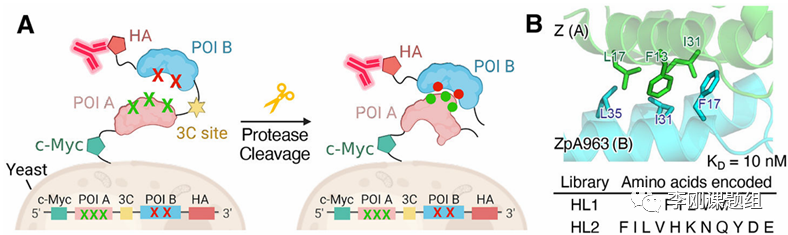
在进化生物学中,共同进化是指在一对生物分子或生物体之间的选择性压力下发生的相互变化,从而适应大自然的选择压力。类推到相互作用的蛋白质之间,也会发生一些耦合突变,以完善其在功能上的相互作用。但是,蛋白质之间的共同进化在实验室一直很难开展。虽然目前定向进化可以筛选出对某种蛋白结合力高的蛋白,但是“文库-文库”的筛选一直充满挑战。另一方面,如何构建一个庞大的共同突变文库并且从中分离出相互作用对,依然是个难题。
这篇文章利用酵母表面展示,构建了“文库-文库”筛选平台,实现了蛋白质的共同进化。然后利用蛋白质语言模型,可以预测一些文库中未编码的氨基酸的相互作用。文章利用酵母EBY100菌株,利用了一对结合力很强的蛋白来测试该模型的效果。将结合界面的6个关键氨基酸进行突变,经过磁珠分选和流式分选,文库序列收敛,和初始序列差别很小,证明了该系统的有效性。然后文章利用这个系统,希望增强另一对结合力较弱的蛋白质的结合能力。经过筛选后,文库的序列逐渐收敛,结合能力得到了不同程度的改善,序列和初始序列差异较大。
接下来,文章开始分析共同进化蛋白质序列之间以及蛋白质结构的关系。不同的高亲和力序列组合之间存在着交叉性和正交性,并且随着A蛋白的突变,B蛋白也会出现相应的补偿突变。一种蛋白的氨基酸突变会导致相应部位的结构发生改变,所结合的另一种蛋白也会发生突变,产生的结构变化会使蛋白互作保持协调,同时周围的未突变氨基酸也会在位置上进行调整,以适应这种改变。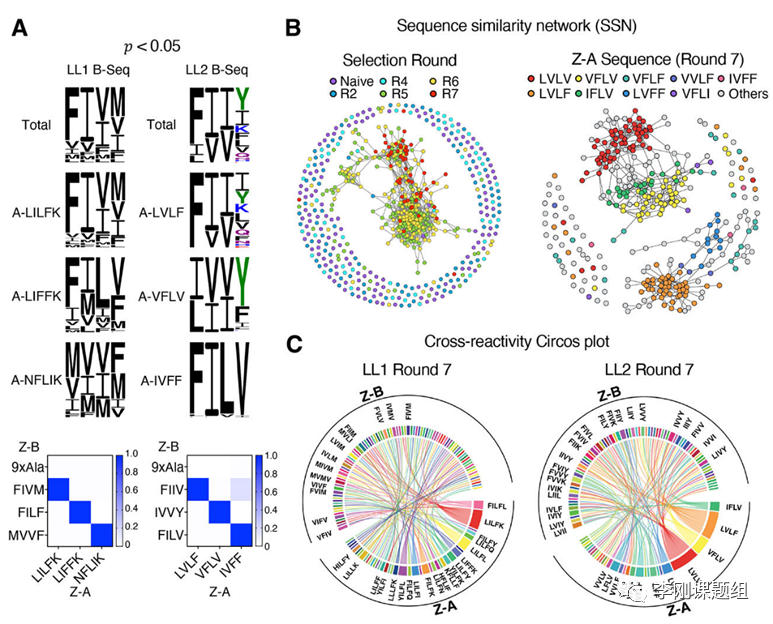
最后文章希望利用蛋白质语言模型以拓展氨基酸种类进而预测更多的相互作用。首先将目前已构建的文库信息嵌入系统,通过迁移学习,对更多氨基酸的文库开展相互作用的预测。将模型的预测结果和实际相比较,发现预测结果和实际结果整体上很相符,对预测结果最好的11个组合开展验证,发现其中6个具有高亲和力。证实了该模型可以在已有的文库基础上拓展氨基酸的范围,从而预测新的相互作用。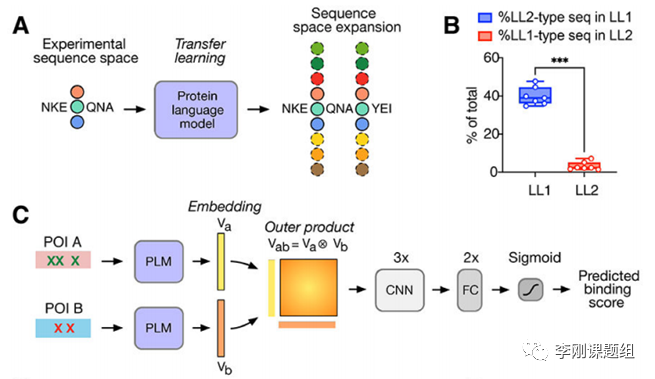
这篇文章利用酵母表面展视,开发了一种蛋白质-蛋白质共同进化的方法,该方法可以改善一对亲和力低的蛋白的相互作用。并且与蛋白质语言模型相结合,可以探索更大的蛋白质相互作用空间,提供了一种生成具有不同分子识别特性的蛋白质复合物的手段,作为生物技术和合成生物学的工具。
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC10403280/#SD1doi.org/10.1126/science.adh1720
感兴趣的读者,可以添加小邦微信(zhiyaobang2020)加入读者实名讨论微信群。添加时请主动注明姓名-企业-职位/岗位 或 姓名-学校-职务/研究方向。